Wir lokalisieren markierte Bakterien in Wurzeln, analysieren Genexpression und Genfunktionen für Kooperation
Forschung
Pflanzen haben sich im Laufe von Millionen von Jahren zusammen mit ihren assoziierten Mikroorganismen entwickelt. Diese Mikroorganismen haben für die Gesundheit, das Wachstum und die Produktivität von Pflanzen eine hohe Bedeutung. Unsere Forschung zielt darauf, diese komplexen Kooperationsmechanismen zu enträtseln und sie für eine nachhaltigere Landwirtschaft zur Anwendung zu bringen.
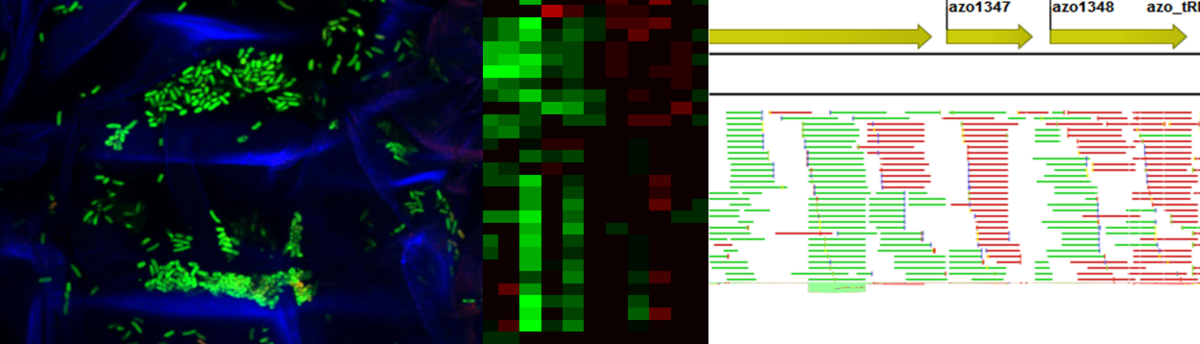
Molekulare Endophyten – Pflanzen-Interaktionen, Azoarcus olearius – Reis Modell
Molekulare Endophyten – Pflanzen-Interaktionen: Azoarcus olearius – Reis Modell
Endophytische Bakterien leben in Pflanzen, ohne krankheits-typische Symptome hervorzurufen. Mehr noch: sie sind nutzbringend für den Wirt. Diese Lebensweise in sehr spezialisierten biologischen Nischen wirft Fragen zur Kooperation auf molekularer Ebene auf: Welche Prozesse ermöglichen die erfolgreiche Etablierung der Mikroorganismen in der Pflanze? Was sind ihre Funktionen im Wirt?
Als Modellsystem für Wurzel-Bakterien-Interaktionen studieren wir Reispflanzen und den Stickstoff-fixierenden Endophyten Azoarcus olearius BH72, der im endophytischen Stadium Nitrogenase-Gene exprimiert.
(Methoden: Genom-und Transkriptom-Analysen wie z.B. RNA-Seq, Mutationsanalysen, synthetische Biologie, Proteom-Analysen, qRT-PCR, Licht-, Fluoreszenz-, Elektronen- und CLSM-Mikroskopie, Reis-Transformation)

Molekulare mikrobielle Ökologie: Diversität und Funktion wurzelassoziierter Bakterien
Reis in situ - Reis im Phytotron und in Testsystemen – Phylogenetischer Stammbaum basierend auf rRNA Genen
Molekulare mikrobielle Ökologie: Diversität und Funktion wurzelassoziierter Bakterien
Unser Wissen über die mikrobielle Vielfalt und Funktion ist immer noch begrenzt. Viele Mikroorganismen widersetzen sich den Kultivierungsansätzen im Labor. Wir versuchen, die funktionelle Biodiversität von Mikroorganismen in Assoziation mit Pflanzen zu analysieren, indem wir Kultivierungsansätze und kultivierungsunabhängige moderne molekulare Werkzeuge kombinieren. Die polyphasische Taxonomie erlaubt es uns, neue Arten zu charakterisieren. Synthetische Gemeinschaften, die aus großen Sammlungen von diversen Pflanzenisolaten bestehen, ermöglichen eine Dekomplexierung des in situ-Systems, um die molekulare Basis der Funktionen des Wurzelmikrobioms besser zu verstehen. Die Analyse der mikrobiellen Gemeinschaft durch Sequenzierung oder Quantifizierung von ribosomalen Markergenen, mRNAs oder Metagenomik ermöglicht es, mehr über die Funktionen in situ zu erfahren, wie in unserem CATCHY-Projekt.
(Methoden: Klassische und neue Hochdurchsatz-Kultivierungstechniken, polyphasische Taxonomie, molekulare Phylogenie, Amplikonsequenzierung Illumina-Platform, p(RT)-PCR).

Rhizobien in Wurzelknöllchen-Symbiose für nachhaltige Landwirtschaft in Afrika
Kuhbohnen Feldversuche - Interaktionen in Subsistenzlandwirtschaft – Probenahme – Aktive Wurzelknöllchen
Rhizobien in Wurzelknöllchen: Symbiose für nachhaltige Landwirtschaft in Afrika
Leguminosen können ihren N-Bedarf durch Wurzelknöllchen-Symbiose mit stickstofffixierenden Rhizobien decken und so Luftstickstoff Pflanzen und Boden zuführen. Eine effiziente Symbiose kann vor allem Kleinbauern in Afrika zugute kommen, indem sie die Produktivität von Hülsenfrüchten steigert, den Boden verbessert und proteinreiche Nahrungsmittel liefert. Um zur Nachhaltigkeit kleinbäuerlicher Anbausysteme in Subsahara-Afrika beizutragen, identifizieren und charakterisieren wir gut angepasste Knöllchen-Symbionten für lokale Hülsenfrüchte wie Kuhbohne, Bambara-Erdnuss, medizinische und undomestizierte Leguminosen (Schwerpunkt Kavango-Region und Nord-Namibia). Viele unserer Isolate zeigen eine hohe Temperaturtoleranz, die für das dortige Klima erforderlich ist. Die Vernetzung mit Stakeholdern, lokalen Partnern und Kleinbauern wird dazu beitragen, die Stickstoff-Fixierung besser zum Einsatz zu bringen. Bisherige Projekte The Future Okavango (TFO) und SASSCAL, derzeit TOPSOIL und SUSTEC.
(Methoden: Kultivierungsansätze, polyphasische Taxonomie, Genomik, Biodünger Produktion im kleinen Maßstab)
