Die Proteine auf der Oberfläche unserer Körperzellen sind mit Zuckern überzogen, die beeinflussen, wie Zellen mit ihrer Umgebung und mit Krankheitserregern interagieren. Sie spielen daher eine wichtige Rolle bei der Erforschung und Herstellung von Medikamenten. Das neu entwickelte, umweltfreundliche und quelloffene Software-Toolkit GlycoSHIELD ermöglicht nun schnelle und effiziente Analysen von Protein-Zuckerschilden. Der neue Ansatz wurde im Rahmen des deutsch-polnischen Dioscuri-Programms der Max-Planck-Gesellschaft in Zusammenarbeit mit der Universität Bremen, der Forschungseinrichtung Inserm in Frankreich und der Academia Sinica in Taiwan entwickelt.
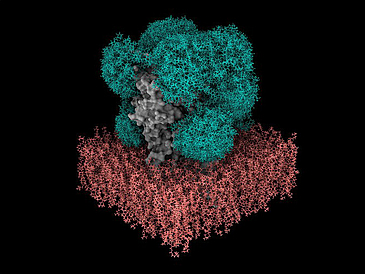
Um zu verstehen, wie Proteine funktionieren und wie sie die Entstehung und den Verlauf von Krankheiten beeinflussen, müssen Forschende ihre dreidimensionale atomare Struktur kennen. Neben experimentellen Methoden nutzen sie dazu auch Computersimulationen. Die künstliche Intelligenz alphafold kann beispielsweise aus der Abfolge der einzelnen Proteinbausteine, den Aminosäuren, zuverlässig die räumliche Struktur eines Proteins voraussagen. Über 75 % der an der Oberfläche unserer Zellen vorhandenen Proteine bestehen jedoch nicht nur aus den Aminosäurebausteinen, sondern sind von Zuckern, so genannten Glykanen, umhüllt. Diese bilden sehr dynamische „Schilde“ um die Proteine. Derzeit ist noch nicht genau bekannt, wie sich diese Schutzschilde verhalten und wie sie die Bindung von Arzneimittelmolekülen beeinflussen. Aufgrund ihrer Mobilität und Variabilität war die Vorhersage der Morphologie von Zuckern, also die Form und Struktur der Zuckermoleküle, bisher schwierig und ressourcenintensiv. Dr. Isabell Louise Grothaus von der Hybrid Materials Interfaces Group unter der Leitung von Professor Lucio Colombi Ciacchi an der Universität Bremen arbeitet seit vier Jahren an Computersimulationen von Glykanen und hat eine Software namens GlyCONFORMER für die Analyse und Klassifizierung ihrer Mobilität und Variabilität entwickelt. Ihr Fachwissen und ihre Software waren ausschlaggebend für die Entwicklung des neuen GlycoSHIELD-Toolkits, das eine schnelle und realistische Modellierung der Zuckerketten auf Proteinoberflächen ermöglicht, wie gerade in der renommierten Zeitschrift Cell berichtet wurde.
Von Tausenden Stunden hin zu wenigen Minuten
Zucker beeinflussen stark, wie Proteine mit anderen Molekülen interagieren. Der Zuckerschild auf dem Spike-Protein des SARS-Cov-2-Coronavirus beispielsweise erschwert die Erkennung des Virus durch Antikörper und verbirgt es so vor unserem Immunsystem. Das zeigt die Relevanz der Zuckerschilde für die Arzneimittelentwicklung – die pharmazeutische Forschung könnte von einer routinemäßigen Vorhersage ihrer Morphologie und Dynamik profitieren. Bislang war die Vorhersage der Struktur von Zuckerschichten mit Hilfe von Computersimulationen jedoch nur mit Fachkenntnissen und speziellen Supercomputern möglich. In vielen Fällen waren dafür Tausende oder gar Millionen von Rechenstunden erforderlich. Mit GlycoSHIELD hat das Team der Wissenschaftler:innen um Dr. Mateusz Sikora, Leiter des Dioscuri Center für die Modellierung von posttranslationalen Modifikationen an der Jagiellonian Universität in Krakau, eine umweltfreundliche Open-Source-Alternative geschaffen. „Unser Ansatz reduziert die Ressourcen, die Rechenzeit und das benötigte technische Know-how“, sagt Sikora. „Jede:r kann nun die Anordnung und Dynamik von Zuckermolekülen auf Proteinen innerhalb weniger Minuten auf seinem PC berechnen.“ Die Software könnte bei der Entwicklung von Medikamenten oder Impfstoffen hilfreich sein, zum Beispiel bei der Immuntherapie gegen Krebszellen, die ein sehr besonderes Profil von Glykanen auf ihrer Oberfläche aufweisen.
Ein Puzzlespiel aus Zucker
Wie ist das Team vorgegangen, um eine so hohe Effizienzsteigerung zu erzielen? Die Forscher:innen erstellten und analysierten eine Bibliothek mit Tausenden von wahrscheinlichen 3D-Positionen der häufigsten Formen von Zuckerketten auf Proteinen, die in Menschen und Mikroorganismen vorkommen. Anhand langer Simulationen und Experimente fanden sie heraus, dass man zuverlässig vorhersagen kann, wie sich bestimmte Glykane verhalten. Es ist dabei zunächst nicht notwendig, die Interaktion zwischen den Hüllen von Zellen oder Teilen der Proteine mit dem angehängten Zucker einzubeziehen. Der Algorithmus basiert auf diesen Erkenntnissen. „GlycoSHIELD-Benutzer:innen müssen nur das Protein und die Stellen, an denen die Zucker gebunden sind, angeben. Unsere Software puzzelt sie dann in der wahrscheinlichsten Anordnung auf die Oberfläche“, erklärt Sikora. „Wir konnten die Zuckerschilde des Spike-Proteins sehr gut reproduzieren, sie sehen genau so aus, wie wir sie in unseren Experimenten sehen!". Mit GlycoSHIELD ist es nun möglich, sowohl neue als auch bestehende Proteinstrukturen mit Zuckerinformationen zu ergänzen. Die Wissenschaftler:innen nutzten GlycoSHIELD darüber hinaus, um das Muster der Zucker auf dem GABAA-Rezeptor, einem wichtigen Ziel für Beruhigungs- und Narkosemittel, aufzudecken.
Weitere Informationen:
Y.-X. Tsai, N.-E. Chang, K. Reuter, H.-T. Chang, T.-J. Yang, S. von Bülow, N. Zerrouki, M. Gecht, C. Penet, I.L. Grothaus, L. Colombi Ciacchi, K.-H. Khoo, G. Hummer, S.-T.D. Hsu; C. Hanus, M. Sikora. Rapid simulation of glycoprotein structures by grafting and steric exclusion of glycan conformer libraries. Cell, DOI: 10.1016/j.cell.2024.01.034 (2024). https://doi.org/10.1016/j.cell.2024.01.034
Über die Bremer Forschungsgruppe: https://www.hmi.uni-bremen.de/index.html
Über die koordinierende Gruppe: https://www.biophys.mpg.de/dioscuri-centre-for-modelling-of-posttranslational-modifications
News des MPI für Biophysik: https://www.biophys.mpg.de/de/news
Fragen beantwortet:
Dr. Isabell Louise Grothaus
Hybrid Materials Interfaces
Bremer Zentrum für Computational Materials Science
Telefon: +49 421 218 64574